
 天天色
天天色
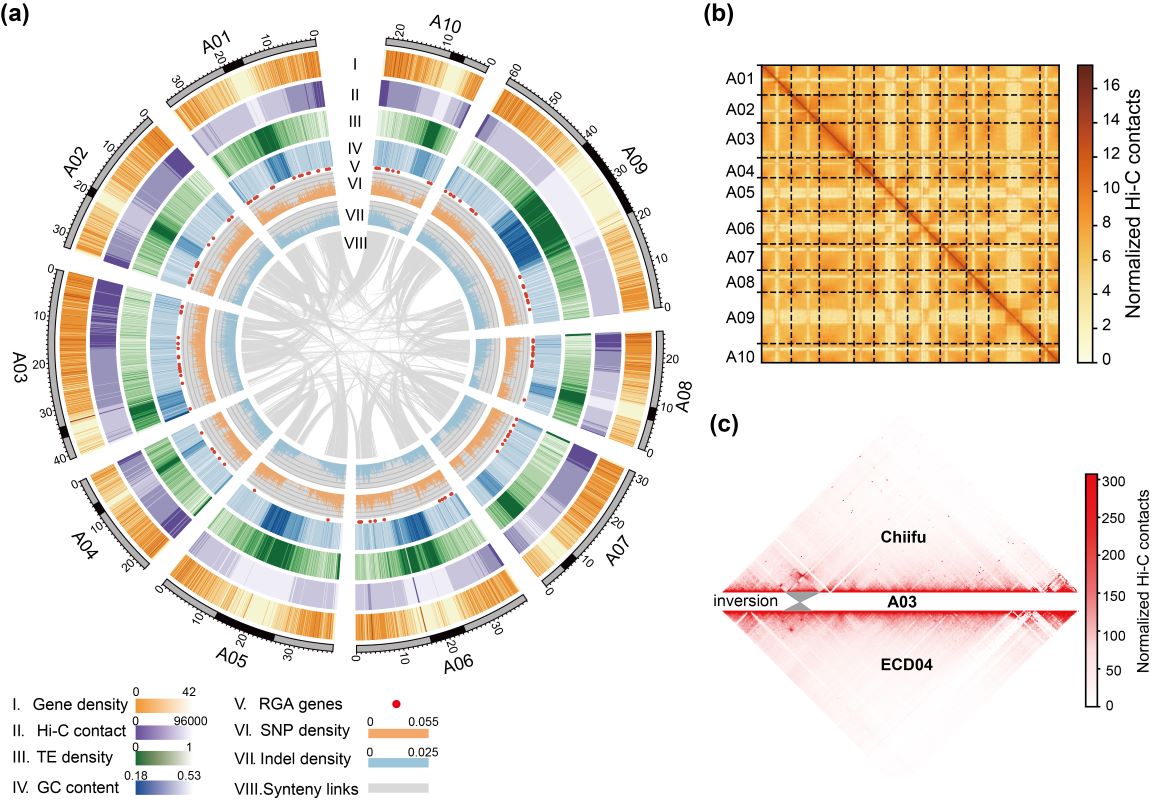
图1 欧洲芜菁ECD04的基因组和Hi-C交互图谱
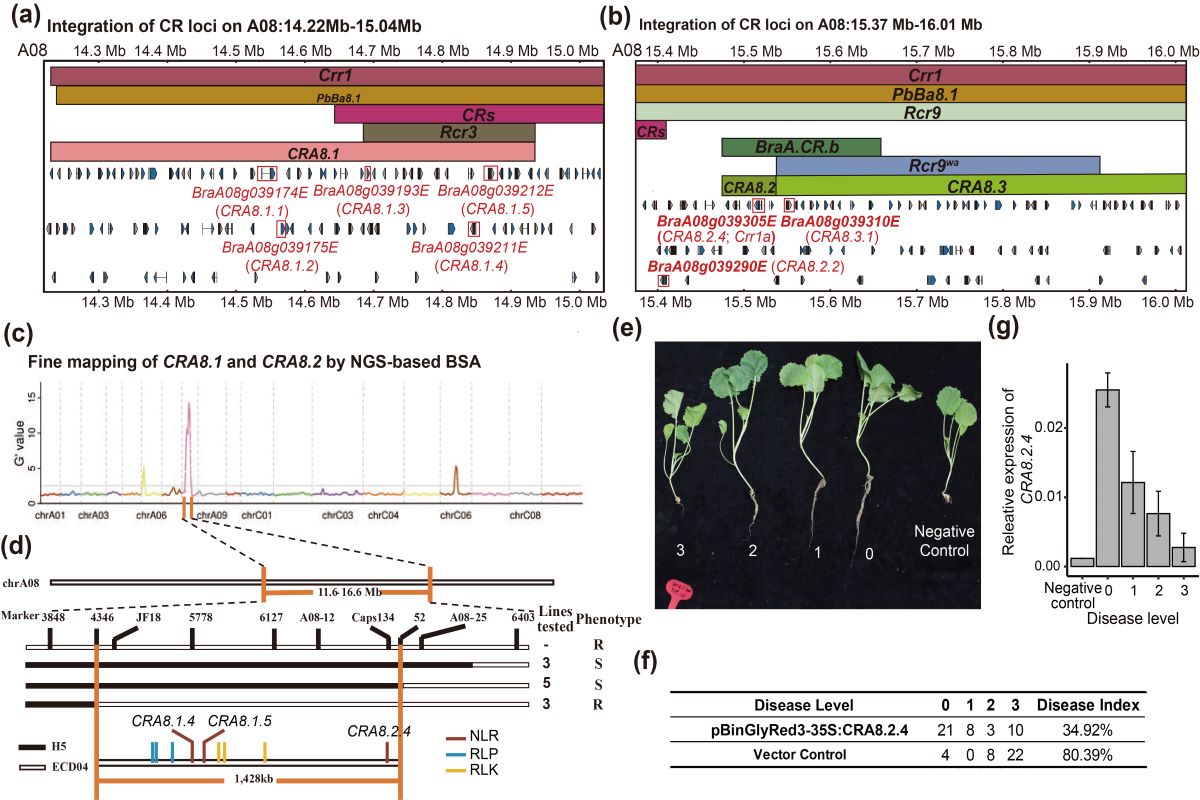
图2 CRA8.2.4位点抗根肿病的功能考证
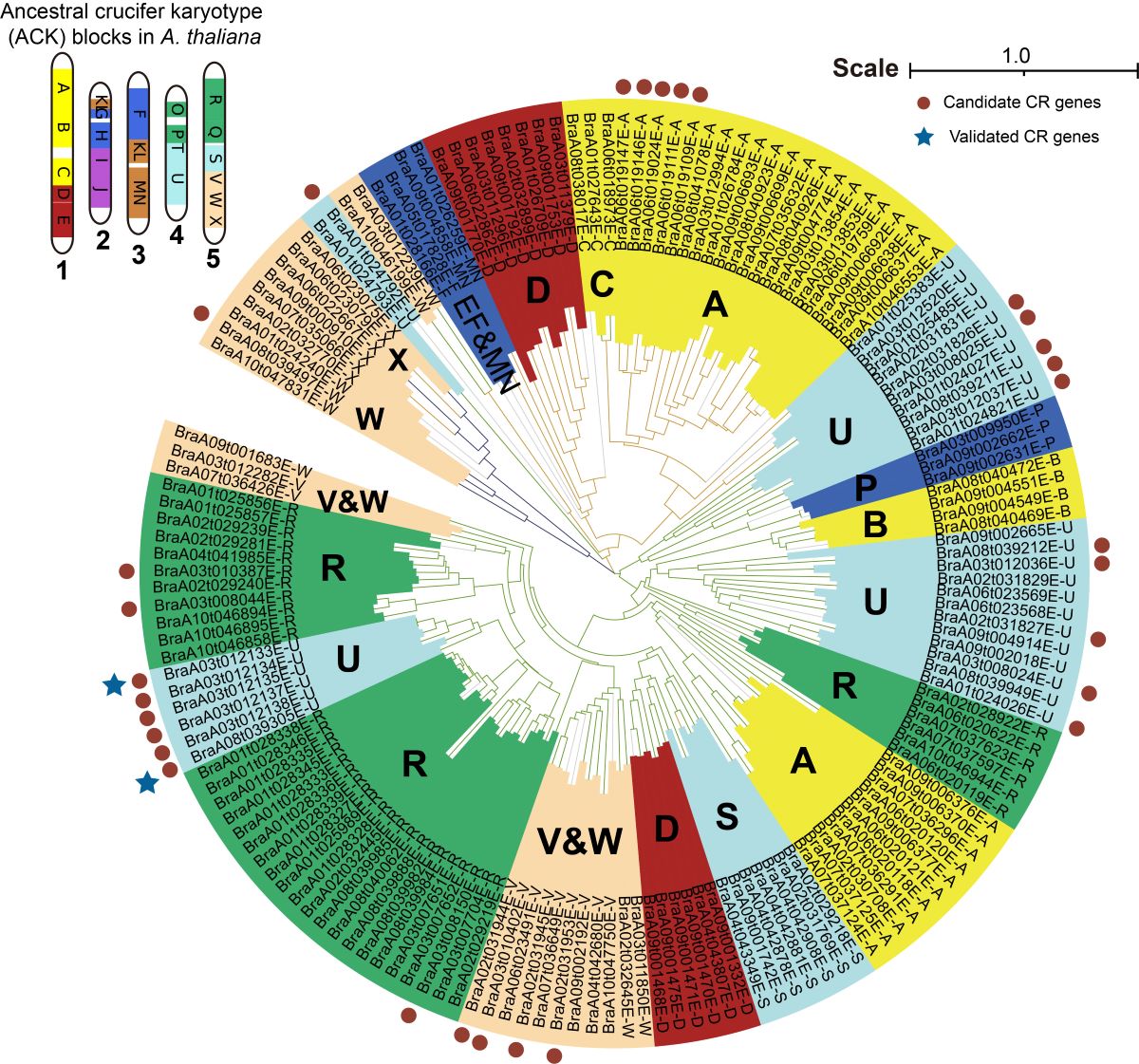
图3 根肿病抗性基因的系统发育树

图4 芸苔属植物进化经过中根肿菌抗性基因的演化模子
南湖新闻网讯(通信员 杨植全 江莹芬)近日,我校油菜团队张椿雨素养、杨庆勇教讲课题组与沈阳农业大学朴钟云素养团队互助征询扫尾以“R gene triplication confers European fodder turnip with improved clubroot resistance”为题在Plant Biotechnology Journal发表。征询愚弄抗根肿病的欧洲芜菁ECD04参考基因组,理解芸薹属根肿病抗性基因的演化历程,为十字花科作物抗根肿病基因的克隆、标识诞生,以及抗性育种提供了蹙迫参考。
根肿病(Clubroot),被称作“十字花科癌症”,是一种由根肿病菌(Plasmodiophora brasicae)引起的一种土传性病害,经常会导致作物根系造成渊博的肿瘤,从而退却作物从泥土中招揽营养与水分,严重者整株作物枯死。十字花科包含好多蹙迫的蔬菜、油料和饲料作物,在我国散布广、面积大,影响额外普通,对富厚安全供给粮油、推动三产交融、助力乡村振兴等方面有蹙迫作用。我国每年约320~400万公顷十字花科作物遭遇根肿病的危害,发病严重的地区赔本可达到60%以上甚而绝收,因此根肿病的防治也曾成为一个蹙迫的计谋问题,从泉源选育根肿病抗性品种被以为是限度根肿病膨胀最灵验的策略之一。现在发现的根肿病抗性基因大多来自芸薹属A基因组,其中大部分的抗性位点起源于欧洲芜菁。但高质料的抗根肿病(Clubroot Resistance,CR)材料的参考基因组的枯竭严重退却了对A基因组中抗性基因的作用机制富厚和育种试验中的挖掘与愚弄。
真實精液大爆射征询东说念主员在征询中遴选了具有多个抗性位点的ECD04抗根肿病欧洲芜菁,承袭第三代PacBio测序时间辘集染色体构象拿获(Hi-C)时间,生效拼装了“ECD04”的基因组,取得了染色体水平的参考基因组序列(图1),并将已报说念的28个抗根肿病位点进行了系统地整合,执意到62个抗根肿病候选基因,并通过转基因和接菌实验考证了两个位点中的候选基因CRA3.7.1和CRA8.2.4对根肿病抗性的功能(图2)。该征询通过相比基因组学和系统发育分析发现多个候选CR基因均来自先人基因组的U区段(图3)。通过与盐芥基因组的共线性分析发现,CRA3.7.1和CRA8.2.4在芸薹属三倍化之前的先人基因组中也曾存在。相比基因组学分析扫尾进一步标明,感病的白菜和油菜基因组的CRA3.7.1和CRA8.2.4序列存在大片断的结构变异以及转座子插入,可能与其抗性的缺失干系。
基于以上扫尾,征询建议芸薹属作物根肿病抗性基因的进化演化模子(图4):全基因组三倍化使得先人基因组中的抗性基因加倍,增强了植株对根肿病的抗性。在芸薹属植物与病菌长久互作和进化经过中,三种互作样子共同存在:1)宿主与病菌达到了动态均衡气象,抗根肿病基因受到浓烈正聘请而一直保留;2)宿主中根肿病抗性冉冉加多,界限了根肿病菌扩散直至冉冉隐藏,最终导致抗根肿病基因功能丧失;3)东说念主类的举止使得芸薹属植株传播到其它莫得根肿病的地区,导致抗性基因功能冉冉丧失。
我校信息学院博士杨植全(现广州大学博士后)和植物科学时间学院博士江莹芬(现安徽省农业科学院作物征询所副征询员)为论文共同第一作家。植物科学时间学院张椿雨素养、信息学院杨庆勇素养和沈阳农业大学园艺学院朴钟云素养为该论文通信作家。作物遗传改变国度重心实验室周永明素养、李再云素养在该征询经过中提供了蹙迫指引和匡助。本面孔得到国度当然科学基金面孔,国度当代农业产业时间体系和中央高校基本科研业务费专项资金等面孔资助。
审核东说念主:杨庆勇
【英文纲领】
Clubroot is one of the most important diseases for many important cruciferous vegetables and oilseed crops worldwide. Different clubroot resistance (CR) loci have been identified from only limited species in Brassica, making it difficult to compare and utilize these loci. European fodder turnip ECD04 is considered as one of the most valuable resources for CR breeding. To explore the genetic and evolutionary basis of CR in ECD04, we sequenced the genome of ECD04 using de novo assembly and identified 978 candidate R genes. Subsequently, the 28 published CR loci were physically mapped to 15 loci in the ECD04 genome, including 62 candidate CR genes. Among them, two CR genes, CRA3.7.1 and CRA8.2.4, were functionally validated. Phylogenetic analysis revealed that CRA3.7.1 and CRA8.2.4 originated from a common ancestor before the whole genome triplication (WGT) event. In clubroot susceptible Brassica species, CR-gene homologs were affected by transposable element (TE) insertion, resulting in the loss of CR function. It can be concluded that the current functional CR genes in Brassica rapa and non-functional CR genes in other Brassica species were derived from a common ancestral gene before WGT. Finally, a hypothesis for CR gene evolution is proposed for further discussion.
论文运动:https://doi.org/10.1111/pbi.13827天天色
